锐谈 | iScience:微生物适应性代谢的模块化升级——发现可扩展磷酸盐分解代谢网络的新型FAD依赖氧化还原酶
- 分类:新闻动态
- 作者:
- 来源:
- 发布时间:2023-12-19 10:52
- 访问量:
锐谈 | iScience:微生物适应性代谢的模块化升级——发现可扩展磷酸盐分解代谢网络的新型FAD依赖氧化还原酶
【概要描述】
- 分类:新闻动态
- 作者:
- 来源:
- 发布时间:2023-12-19 10:52
- 访问量:
详情
背景:
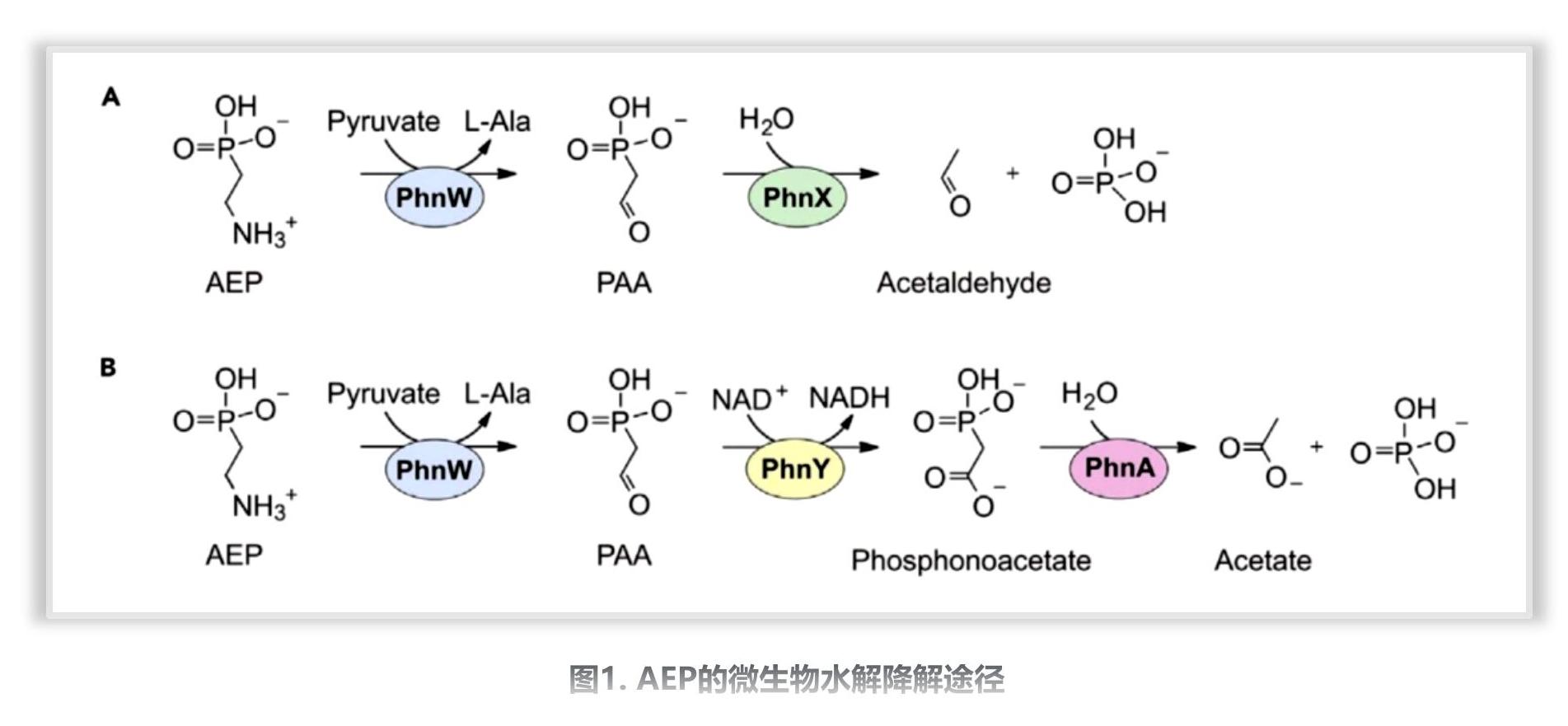
磷酸盐对生物体的生长发育非常重要,但在一些环境中可利用的无机磷源有限。脱碳磷酸盐是一类含有直接的C-P键的有机化合物,在环境中广泛存在。其中2-氨基乙基磷酸(AEP)是最常见的一种天然脱碳磷酸盐。许多细菌具有通过特异的“水解”途径降解AEP的能力,以获取磷源(图1)。

尽管AEP水解途径广泛存在于多个细菌中,但这些途径对底物范围非常狭窄,只能降解AEP本身。考虑到AEP相关化合物N-甲基AEP(M1AEP)也很常见,本研究人员推测一些辅助酶可能会有此活性,因此将M1AEP(N-methyl AEP)也引入AEP水解途径中。

实验部分:
在AEP水解相关基因簇中发现编码FAD依赖氧化还原酶的基因非常常见。这些酶至少可以归于PbfB、PbfC和PbfD三个亚组。重组表达并纯化了来自不同亚组的代表性酶后进行表征,发现这些酶都可以将M1AEP氧化为PAA(图2)。


总结与讨论:
1. FAD酶扩大了AEP水解途径的应用范围,这些酶允许细菌利用AEP相关化合物,提高对多种营养源的适应性。
2. 不同FAD酶功能的差异适应不同细菌需求,如PbfC酶高专一性适合不需要降解AEP的细菌;PbfD酶适合缺少PhnW的菌种。
3. FAD酶基因的水平传递,这些辅助酶编码基因可能与AEP水解核心基因一起在细菌之间传递,组装成适应不同环境的代谢模块。
4. 对AEP微生物代谢网络的新认识,AEP水解途径表现出高度的模块化和可重组性,可根据环境需求组合生成不同途径。

作者:迪迦

关键词:
搜索


×
搜索
版权所有 © 2022 太阳集团tyc5997